肿瘤特异性表达的体细胞突变、可变剪切、融合基因、非编码RNA、环状RNA等均有可能产生肿瘤特异性抗原多肽(neoantigen)【1-5】。这些肽段与MHC结合并经过抗原呈递,最终有可能产生免疫反应,从而在肿瘤的免疫治疗中具有重要的作用。近年来,基于来自肿瘤样本的基因组数据进行肿瘤特异性抗原的预测已成为一种常用的肿瘤特异性抗原发现方法。这种方法通常产生大量的候选肽段且具有很高的假阳性。高精度质谱测序数据能够为抗原多肽对应的突变在蛋白水平的表达提供证据,从而进一步缩小肿瘤特异性抗原筛选的范围并降低假阳性。
近日,美国贝勒医学院章冰教授团队在Nature Communications杂志上发表方法研究型长文:Cancer neoantigen prioritization through sensitive and reliable proteogenomics analysis,报道了一套新的基于多组学(proteogenomics)的肿瘤特异性抗原预测流程NeoFlow。
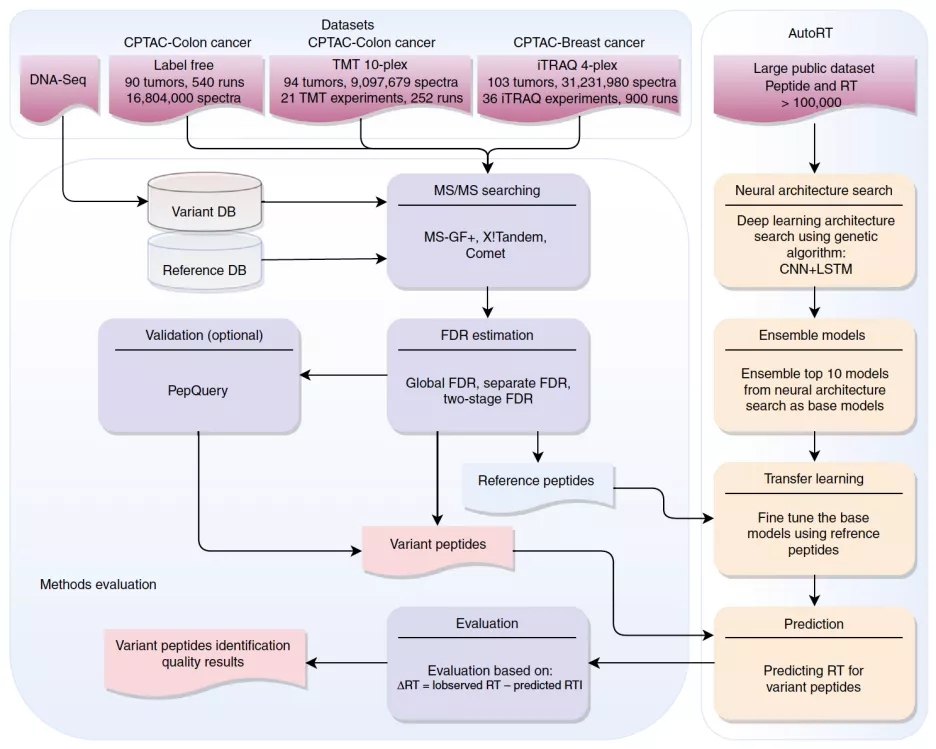
液相色谱-串联质谱(LC-M/MS)联用技术是目前进行大规模肽段/蛋白检测的主流技术。该技术可以和基因组测序技术结合使用来检测新肽段, 比如肿瘤特异性抗原多肽。在这项研究中,研究人员首先对基于LC-MS/MS产生的质谱数据的常用的不同的新肽段检测质控方法进行评估(图1)。为了系统的评估不同方法对新肽段的检测效果,研究人员开发了一套基于自动化深度学习并结合迁移学习的肽段保留时间预测算法AutoRT(图1)。
图 1实验总体设计。
肽段保留时间(retention time)是指肽段经过液相色谱系统(LC)分离进入质谱系统(MS)所需的时间,它主要是由肽段的理化性质和LC条件决定的。在常用的基于质谱数据的肽段检测方法里,肽段保留时间在评估肽段鉴定可信度过程中并没有用到。而通常来讲,如果精确预测的肽段保留时间与实际质谱记录的保留时间差别较小,那么该肽段鉴定的可信度就较高,反之就较低。因此该研究将肽段预测的保留时间与实际观测到的保留时间的差值用作一个相对独立且无偏好的肽段鉴定可信度的度量指标。基于此,研究人员利用新开发的AutoRT来对不同的新肽段检测方法在三个来自CPTAC的大规模肿瘤蛋白质组数据集上进行系统评估(图1)。
通过与其它基于传统机器学习算法的保留时间预测软件以及基于深度学习的保留时间预测软件相比,AutoRT在所有测试数据上均表现出了最好的预测性能。在对来自3个CPTAC数据集的多达1600多个实验数据的测试表明,AutoRT的肽段保留时间预测的准确性(MAE <= 0.7 minute)达到了能够用于肽段鉴定可信度评估的要求。
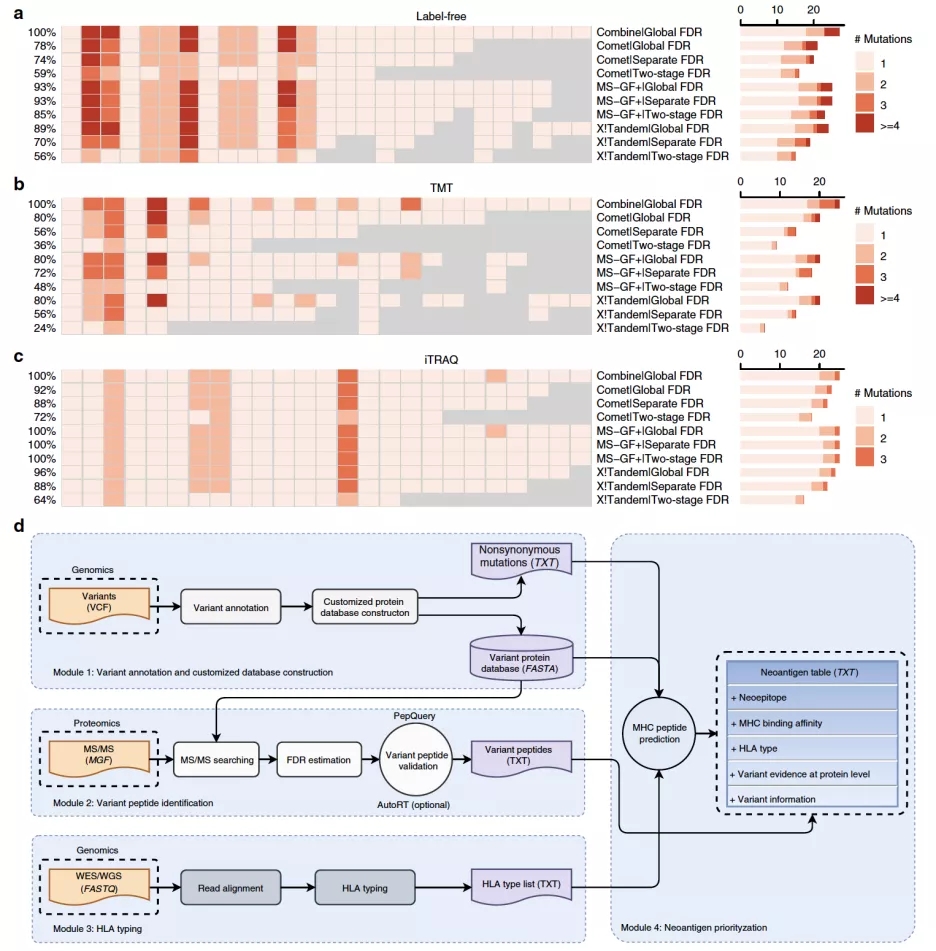
在本研究中,研究人员利用匹配的肿瘤样本的DNA测序数据来构建包含来自体细胞突变(somatic mutation)和生殖细胞突变(germline mutation)的肽段的个性化蛋白数据库用于质谱数据的分析。并进一步对有质谱证据支持的somatic mutation进行neoantigen的预测。通过利用AutoRT对不同新肽段检测方法在三个CPTAC数据集上的的系统评估表明:(1)在同一数据集上,使用不同新肽段检测FDR质控方法鉴定到的新肽段数量上差别很大;(2)PepQuery【6】(章冰教授团队之前开发的用于新肽段的鉴定和验证软件,已发表于Genome Research上)与不同FDR质控方法结合均能有效的去除假阳性鉴定;(3)在保证总体鉴定质量的同时,全局水平FDR控制(Global FDR)结合PepQuery验证具有最高的灵敏度;(4)结合多个肽段鉴定搜索引擎的结果,能进一步提高neoantigen的鉴定灵敏度(图2)。
图 2不同方法neoantigen预测结果的比较以及NeoFlow流程。
最终研究人员根据对不同方法的系统评估结果开发了一套新的整合基因组数据和蛋白质组数据用于肿瘤特异性抗原多肽的预测流程NeoFlow(图2)。NeoFlow包含四个功能模块:(1)变异注释和样本个性化蛋白数据库构建;(2)基于质谱数据的新肽段鉴定;(3)基于全基因组测序(WGS)数据或者全外显子组测序(WES)数据的HLA类型预测;(4)Neoantigen预测。这套流程也能用于免疫多肽组(immunopeptidomics)数据的分析。
原文链接:
https://www.nature.com/articles/s41467-020-15456-w
参考文献
1. Zhang, M. et al. RNA editing derived epitopes function as cancer antigens to elicit immune responses. Nat. Commun. 9, 3919 (2018).
2. Ott, P. A. et al. An immunogenic personal neoantigen vaccine for patients with melanoma. Nature 547, 217–221 (2017).
3. Sahin, U. et al. Personalized RNA mutanome vaccines mobilize poly-specific therapeutic immunity against cancer. Nature 547, 222–226 (2017).
4. Smith, C.C., Selitsky, S.R., Chai, S. et al. Alternative tumour-specific antigens. Nat Rev Cancer 19, 465–478 (2019).
5. Yang, W., Lee, K., Srivastava, R.M. et al. Immunogenic neoantigens derived from gene fusions stimulate T cell responses. Nat Med 25, 767–775 (2019).
6. Wen, B., Wang, X. & Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29, 485–493 (2019).
扫描上面二维码在移动端打开阅读